前段时间,实验室的一瓶未使用过的 YPD 培养基发现染菌了,出现了一大朵类似蘑菇的真菌,形态奇异。想着好好的培养基不能白白浪费了,要让这偷吃的真菌付出代价,于是小新决定提取这个未知真菌DNA。(另外,对这种真菌有所了解的小伙伴可以在评论区留言,让小新知道这个可恶的家伙叫什么。)
实验目的
提取未知真菌的 DNA。
实验材料
1.未知真菌样本;
3.电子恒温不锈钢水浴锅(上海宜昌仪器纱筛厂)
4.旋涡振荡器(越新仪器,XH-C)
5.台式离心机(eppendorf Centrifuge 5415 D)
7.电泳仪(北京六一仪器厂,DYY-6C 型)
实验步骤及结果
(一)DNA提取环节
众所周知,真菌样本因为多糖多酚的特性,核酸较难提取,所以本次实验使用植物/真菌 DNA 试剂盒提取未知真菌样本的 DNA,具体操作步骤如下:
1.称取 1500 mg 真菌于研钵中,加入 600 μl 65℃预热的 Buffer PD 和 6 μl β-巯基乙醇,用力研磨至匀浆状。
2.研磨充分后加入 2400 μl 65℃预热的 Buffer PD,继续研磨 1 分钟,使组织完全裂解。(研磨到最后,研钵中还是存在较多细小颗粒状物质,无法被彻底研磨成匀浆。
3.转移 800 μl 裂解产物至两个 2 ml 离心管中,将离心管置于 65℃水浴 30 分钟。水浴期间每隔 5~10 分钟翻转离心管数次以帮助 DNA 的释放。
4.加入 800 μl Buffer EX,用力混合均匀,12000 rpm 离心 5 分钟。
5.小心吸取 600 μl 上清,转入一个新的 1.5 ml 管中。
6.在上清中加入等体积的 Buffer GP,混合均匀。
7.吸取 600 μl 步骤 6 中的混合液加入到核酸纯化柱中,12000 rpm 离心 30 秒弃滤液。
8.将步骤 6 中剩余的混合液全部加入到核酸纯化柱中,12000 rpm 离心 30 秒弃滤液。
9.在核酸纯化柱中加入 500 μl Buffer WA,12000 rpm 离心 30 秒弃滤液。
10. 在核酸纯化柱中加入 600 μl Buffer WB,12000 rpm 离心 30 秒弃滤液。
11. 14000 rpm 空离 1 分钟。
12. 将核酸纯化柱置于一个洁净的 1.5 ml 离心管中,在纯化柱中加入 200 μl 65℃预热的 Buffer TE,盖上管盖,室温静置 2 分钟,12000 rpm 离心 1 分钟,洗脱得到 DNA。
13. 在超微量分光光度计上用 Buffer TE 调零,测量提取好的 DNA,结果如下:
表一:未知真菌提取的 DNA 浓度
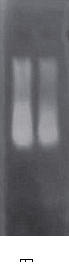
14.在 1%的琼脂糖凝胶上,加入 5 μl 提取到的 DNA,电泳 15 分钟,结果下图所示:
图一
从浓度测量结果分析可知,植物/真菌 DNA 提取试剂盒成功提取出了未知真菌的 DNA,且浓度颇高。但是 A260/A280 比值达到 2.00,推测提取的 DNA 里面含有大量 RNA 或者降解的 DNA。经电泳图验证,确实存在大量拖带,与测量所得数据相符合。
(二)DNA 处理纯化环节
为了验证拖带是 RNA 还是降解的 DNA,往提取的 DNA 中加入 0.5 μl RNaseA,37℃放置 1 小时,然后再用 DNA 纯化试剂盒提取处理过的 DNA,具体步骤如下:
1.吸取 30 μl RNase A 处理过的 DNA 溶液,加入 5 倍体积的 Buffer P,直接用移液器吸打几次混匀,将混合液转移到核酸纯化柱中。
2.12000 rpm 离心弃滤液。
3.在核酸纯化柱中加入 700 μl Buffer WB,12000 rpm 离心弃滤液。
4.14000 rpm 空离 1 分钟。
5.将核酸纯化柱置于一个洁净的 1.5 ml 离心管中,在纯化柱的膜中央加入 30 μlBuffer TE,盖上管盖,室温静置 1 分钟,12000 rpm 离心 30 秒洗脱 DNA。
6.在超微量分光光度计上用 Buffer TE 调零,测量提取好的 DNA,结果如下:
表二:经 RNase A 处理过的未知真菌的 DNA 浓度
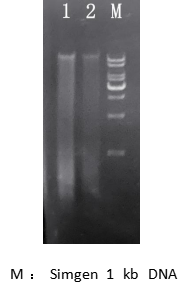
7.在 1%的琼脂糖凝胶上,加入 5 μl 提取到的 DNA 电泳 25 分钟,结果如下图:
图二
对比表二和表一数据可以看到,经过RNase A 处理后 DNA 溶液的浓度减少了 10 倍以上,说明之前提取的 DNA 中含有大量的 RNA。从电泳图可以看到,经过 RNase A 处理后,拖带变少了,这是因为其中的大量 RNA 被降解去除了。但是拖带情况仍旧存在,说明其中含有一部分降解的 DNA。
讨论与分析
1.本次实验成功提取到了未知真菌样本的 DNA,从图二可以明显看到,虽然有部分降解,但提取的 DNA 主带清晰可见。
2.对比样本 1 和样本 2 的数据可以看到,两管样本的 DNA 浓度相差了 2 倍左右,推测可能是因为研磨时该真菌样本中存在较多的颗粒物质,在提取步骤3 转移到离心管时,第一个样本吸取的大多是上清液,第 2 个样本吸取了较多底部的这种颗粒,使得两管样本最后提取的 DNA 数据相差较大。
3.从表一和表二数据分析可知,该未知真菌提取的 DNA 中含有大量的 RNA,占了总浓度的 90%以上,相对来说 DNA 含量比较少,平均 500 mg 样本中只有约 5.7 μg DNA。这也是真菌样本(比如酵母)的共通点:DNA 含量较少而RNA 含量高,因此建议大家在提取真菌时尽可能的多加样本,确保能提到足够的 DNA,并且在提取步骤 3 中补加 RNase A 储存液(Simgen Cat. No. 8001001)用以消化 RNA。
众所周知,DNA 所包含的遗传信息决定了不同种生物之间的差异,真菌也不例外。既然我们提取到了它的 DNA,就有可能通过 DNA 分子来鉴定它到底是哪一类真菌,具体方法和结果敬请期待。