还记得前段时间偷吃实验室培养基的真菌吗?之前小新用植物/真菌DNA试剂盒(Simgen Cat.No.3200050)提取了该真菌的基因组DNA。这次小新用提取的DNA进行了PCR扩增,然后将PCR产物送去基因测序,最后在NCBI上与已知菌种序列进行比对后得出该菌种为曲霉属(Aspergillus sp.)。下面就跟小新一起回顾一下具体的操作步骤吧。
实验目的:
鉴定未知真菌菌种。
实验材料:
1. 样本:提取好的真菌DNA。
2. 2×PCR Mix(Simgen Cat.No.7003100)、真菌ITS引物(ITS1:5?-TCCGTAGGTGAACCTGCGG-3’/ITS4:5?-TCCTCCGCTTATTGATATGC-3?)和(ITS5:5?-GGAAGTAAAAGTCGTAACAAGG-3?/ITS4:5?-TCCTCCGCTTATTGATATGC-3?)
3. PCR仪:Techne FPROGO5Y Progene Thermal
4. 电泳仪(北京六一仪器厂,DYY-6C型 )
5. 相关网址:https://www.ncbi.nlm.nih.gov/
实验内容:
1. PCR扩增:
① 将2×PCR Mix、ddH2O、模板DNA和引物室温解冻,置于冰上。
② 将解冻后各个组分上下翻转混合均匀,按下列组成配制PCR反应液:

③ 手指轻弹PCR反应管充分混匀,简短离心。
④ 按下方条件设置PCR程序:
94℃,4 min→(94℃,45 s→55℃,45 s→72℃,1 min)×35 cycles→72℃,5 min
2. 对PCR产物进行琼脂糖凝胶电泳检测。
3. 将获得的PCR扩增产物及引物交由测序公司测序。
4. 在NCBI网站上对比序列,确定真菌菌种。
打开NCBI,选择BLAST,选择Nucleotide Blast,在Enter Query Sequence序列框内输入核苷酸序列,输入Job Title,在默认条件下BLAST,得到结果。
5. MEGA5碱基序列比对。
打开MEGA5,将菌种的碱基序列输入,点击W进行比对,删除两端不能比对的碱基序列,将比对结果保存。关闭页面,在主页面打开Analysis→Phylogeny→Construct/Test Neighbor-Joining Tree...参数选择Bootstrap method输入1000,开始建树。
实验结果
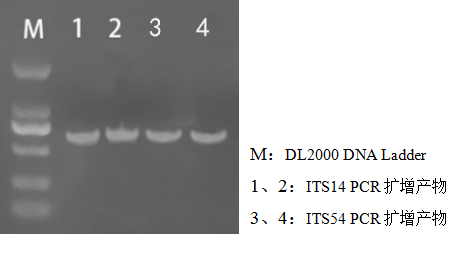
1. 凝胶电泳结果:
在1%的琼脂糖凝胶上,加入5 μl扩增产物、DL2000 DNA Ladder,电泳15分钟,结果如下:
2. 序列比对结果:
NCBI Nucleotide BLAST结果截图(部分)如下:


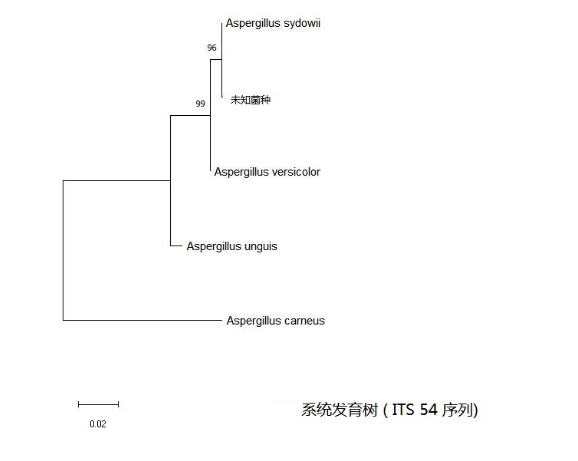
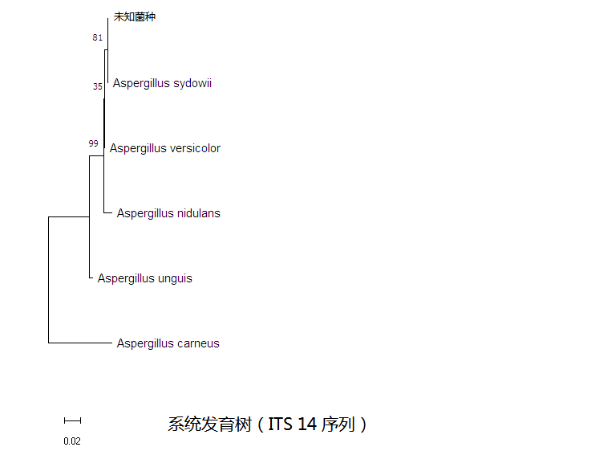
通过MEGA5软件对未知菌种及其相似菌株核苷酸序列进行比
对,删除两侧多余的不能比对的序列,对未知菌种及其相似菌株核苷酸序列分别建立系统发育树,结果如下:
讨论与分析
1. 本次真菌鉴定是通过扩增并测序未知菌种的ITS序列,然后与已知真菌ITS序列比较,从而获得未知真菌的种属信息。为了提高准确率,使用了两对引物分别进行扩增、测序和对比。
2. 从电泳图能看到,两对引物均扩增出了条带,且位置差不多,序列对比结果也几乎一致。根据NCBI BLAST的结果来看,未知菌种应属于曲霉属(Aspergillus sp.)。其菌种核苷酸序列与Aspergillus sydowii、Aspergillus versicolor等曲霉属的菌种核苷酸序列比对结果相似度都很高。
综上,最终确定了这个偷吃了实验室培养基的真菌是一种曲霉菌。